Otro enfoque es el someter a las características predictivas a pruebas estadísticas univariadas y seleccionando las que mejores resultados devuelvan. Por ejemplo, la clase SelectKBest requiere ser instanciada indicando el test estadístico a aplicar y el número de características a seleccionar. Los test disponibles dependen del objetivo del análisis:
Regresión: r_regression, f_regression, mutual_info_regression
Clasificación: chi2, f_classif, mutual_info_classif
Por ejemplo, el test chi-cuadrado (X2) mide la dependencia entre cada una de las características predictivas y la variable objetivo. Si el valor devuelto es pequeño, significa que ambas variables están poco relacionadas, mientras que un valor grande supone una fuerte asociación entre ellas. En este caso, la significancia estadística se determina comparando el valor obtenido con un valor crítico.
Apliquemos este método al dataset wine (que suponemos ya cargado):
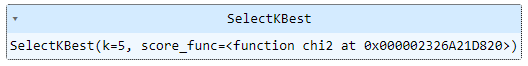
Importamos e intanciamos la clase SelectKBest especificando el test a realizar (chi-cuadrado) y el número de características a extraer (5, por ejemplo):
Ahora podríamos entrenar el selector (usando el método .fit()), o entrenar y transformar las características predictivas (usando el método .fit_transform()). Entrenemos simplemente el selector:
Podemos visualizar el resultado del test para cada una de las características usando el atributo .scores_ (antes de ello, vamos a fijar un máximo de 2 cifras decimales al mostrar arrays NumPy para facilitar la lectura de los valores):
10.52, 0.53, 1.33, 45.8 , 0. , 1.51,
14497.07])
Recordemos que un valor mayor implica una asociación más fuerte con la variable objetivo.
Los p-values correspondientes están accesibles a través del atributo .pvalues_:
0.22, 0. ])
El método .get_support() nos devuelve un array de booleanos indicando si la característica predictiva correspondiente superó o no el umbral:
True, False, False, True])
Por último, el método .get_feature_names_out() devuelve los nombres de las N características seleccionadas:
'proline'], dtype=object)