Aunque solo sea como ejercicio, apliquemos esta técnica para transformar el dataset Iris realizanco los cálculos manualmente. Los pasos a seguir son:
- Obtención de la matriz de covarianza de las características
- Obtención de los eigenvectors (representando los componentes principales)
- Ordenación de éstos según sus eigenvalues
- Selección de los N componentes principales correspondientes a las N dimensiones del espacio al que vamos a transformar los datos
Comenzamos cargando los datos:
data.head()
Y extraemos las características predictivas:
PCA es muy sensible a la escala de las características, por lo que, si deseamos dar a todas ellas la misma importancia, convendrá escalarlas adecuadamente:
X = scaler.fit_transform(X)
Calculamos ahora la matriz de covarianza de las características predictivas transformadas:
cov_matrix
[-0.11835884, 1.00671141, -0.43131554, -0.36858315],
[ 0.87760447, -0.43131554, 1.00671141, 0.96932762],
[ 0.82343066, -0.36858315, 0.96932762, 1.00671141]])
Podemos obtener los eigenvectors (vectores propios) y eigenvalues (valores propios) de la matriz de covarianza usando la función np.linelg.eig() de NumPy:
Los vectores propios (vectores que, en general, al ser transformados solo van a ver modificado su módulo, no su dirección) son los siguientes:
[-0.26934744, -0.92329566, 0.24438178, -0.12350962],
[ 0.5804131 , -0.02449161, 0.14212637, -0.80144925],
[ 0.56485654, -0.06694199, 0.63427274, 0.52359713]])
Los vectores propios están situados en columnas. Ésta es la matriz que va a transformar los datos desde el espacio original hasta el nuevo espacio.
Los eigenvalues (escalares por los que se multiplicarán los vectores propios cuando sean transformados y que representan la varianza explicada por el componente principal correspondiente) son los siguientes:
Estos valores nos permiten estimar el porcentaje de varianza de las características originales explicada por cada componente principal, simplemente dividiendo cada valor por la suma de los cuatro valores:
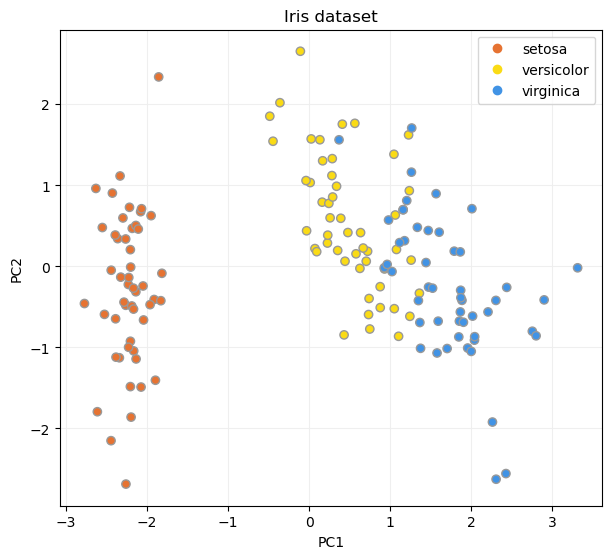
Vemos que, en el dataset Iris, el primer componente principal supone el 72.9% de la varianza total.
Ordenamos los vectores propios según sus valores propios, en orden decreciente:
eig_values = eig_values[idx]
eig_vectors = eig_vectors[:, idx]
[-0.26934744, -0.92329566, 0.24438178, -0.12350962],
[ 0.5804131 , -0.02449161, 0.14212637, -0.80144925],
[ 0.56485654, -0.06694199, 0.63427274, 0.52359713]])
(en nuestro caso la matriz no ha cambiado)
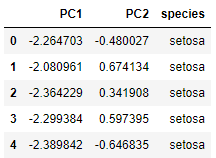
Y, ahora, podríamos seleccionar solo los dos primeros componentes principales, reduciendo, de esta forma, nuestro dataset a dos características:
X_pca = X.dot(W)
[-2.08096115, 0.67413356],
[-2.36422905, 0.34190802],
[-2.29938422, 0.59739451],
[-2.38984217, -0.64683538]])
Reconstruyamos el DataFrame con estas características:
data_pca = pd.concat([data_pca, data['species']], axis = 1)
data_pca.head()
Y mostremos el resultado en un diagrama de dispersión:
ax.set_aspect("equal")
scatter = ax.scatter(
x = data_pca.PC1, y = data_pca.PC2,
c = data_pca.species.astype("category").cat.codes,
zorder = 2, edgecolor = "#999999",
cmap = ListedColormap(["#E67332", "#FADB15", "#4193E5"])
)
ax.set_title("Iris dataset")
ax.set_xlabel("PC1")
ax.set_ylabel("PC2")
ax.legend(
handles = scatter.legend_elements()[0],
labels = list(data_pca.species.unique())
)
ax.grid(color = "#EEEEEE", zorder = 1, alpha = 0.9)
plt.show()