Visualicemos cuáles serían las fronteras de decisión para el modelo entrenado a partir de dos características predictivas (sepal_length y sepal_width, por ejemplo), y filtremos el dataset para que solo incluya las mencionadas clases versicolor y virginica:
iris = iris[iris.species.isin(["versicolor", "virginica"])]
iris["label"] = iris.species.astype("category").cat.codes
y = iris.label
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state = 0)
from sklearn.linear_model import LogisticRegression
model = LogisticRegression(solver = 'lbfgs')
model.fit(X_train.values, y_train.values)
Apliquemos ahora nuestra función show_boundaries al modelo resultante:
show_boundaries(model, X_train.values, X_test.values, y_train, y_test, iris.species.unique())
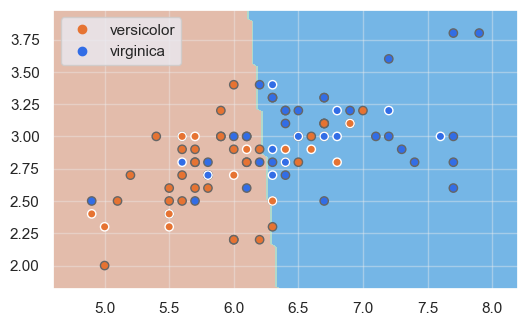
Recordemos que, en esta imagen, se muestran con borde oscuro las muestras del conjunto de entrenamiento, y con bordes blancos las del conjunto de validación.
Tal y como cabía prever, al tratarse de un conjunto de datos no linealmente separable, hay un cierto porcentaje de muestras del dataset original que no son correctamente clasificadas.