Probemos a realizar el mismo ejercicio con dos clases linealmente separables, como las especies setosa y virginica:
iris = sns.load_dataset("iris")
iris = iris[iris.species.isin(["setosa", "virginica"])]
iris["label"] = iris.species.astype("category").cat.codes
iris = iris[iris.species.isin(["setosa", "virginica"])]
iris["label"] = iris.species.astype("category").cat.codes
X = iris[["sepal_length", "sepal_width"]]
y = iris.label
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state = 0)
from sklearn.linear_model import LogisticRegression
model = LogisticRegression(solver = 'lbfgs')
model.fit(X_train.values, y_train.values)
y = iris.label
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state = 0)
from sklearn.linear_model import LogisticRegression
model = LogisticRegression(solver = 'lbfgs')
model.fit(X_train.values, y_train.values)
Mostramos las fronteras de decisión:
show_boundaries(model, X_train.values, X_test.values, y_train, y_test, iris.species.unique())
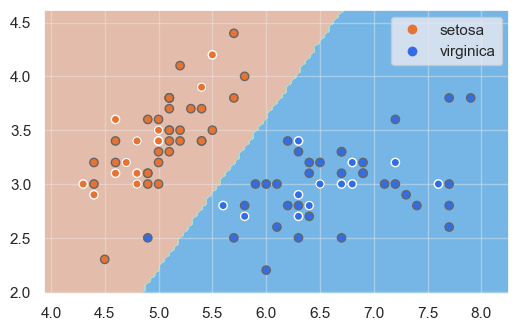
En este caso vemos que una muestra del dataset de entrenamiento no ha sido correctamente clasificada. Recordemos, en todo caso, que hay un cierto número de hiperparámetros que podríamos modificar, como el número de iteraciones a aplicar en el algoritmo de descenso de gradiente, el optimizador, etc.