Entrenemos el modelo con el dataset de entrenamiento y veamos el comportamiento que ha tenido si lo aplicamos al dataset de validación. Eso sí, la tasa por defecto en el Perceptrón proveído por Scikit-Learn es de 1, valor demasiado grande si tenemos en cuenta que hemos escalado los datos, de forma que especifiquemos un valor menor:
model.fit(X_train_std, y_train)
El porcentaje de muestras bien clasificadas en el dataset de validación es:
Un 71%. Y el número de epochs necesarias antes de que haya terminado el entrenamiento ha sido:
Los pesos para las dos entradas de cada uno de los tres modelos que se han entrenado (recordemos que se está aplicando la técnica de one vs. rest son:
[-0.05461475, -0.1817912 ],
[ 0.24889318, -0.00474238]])
y los bias:
Podemos mostrar en sendas gráficas las fronteras de decisión situando los datos de entrenamiento y los datos de validación:
fig, ax = plt.subplots(1, 2, figsize = (10, 6))
ax[0].set_aspect("equal")
ax[1].set_aspect("equal")
plot_decision_boundaries(model, X_train_std, ax[0])
plot_decision_boundaries(model, X_train_std, ax[1])
scatter = ax[0].scatter(
x = X_train_std[:, 0], y = X_train_std[:, 1], c = y_train,
cmap = colors, zorder = 2, edgecolor = "#666666")
scatter = ax[1].scatter(
x = X_test_std[:, 0], y = X_test_std[:, 1], c = y_test,
cmap = colors, zorder = 2, edgecolor = "#666666")
ax[0].legend(
handles = scatter.legend_elements()[0],
labels = list(iris.species.unique())
)
ax[1].legend(
handles = scatter.legend_elements()[0],
labels = list(iris.species.unique())
)
ax[0].grid(color = "#EEEEEE", zorder = 1, alpha = 0.4)
ax[1].grid(color = "#EEEEEE", zorder = 1, alpha = 0.4)
ax[0].set_title("Dataset de entrenamiento")
ax[1].set_title("Dataset de validación")
plt.tight_layout()
plt.show()
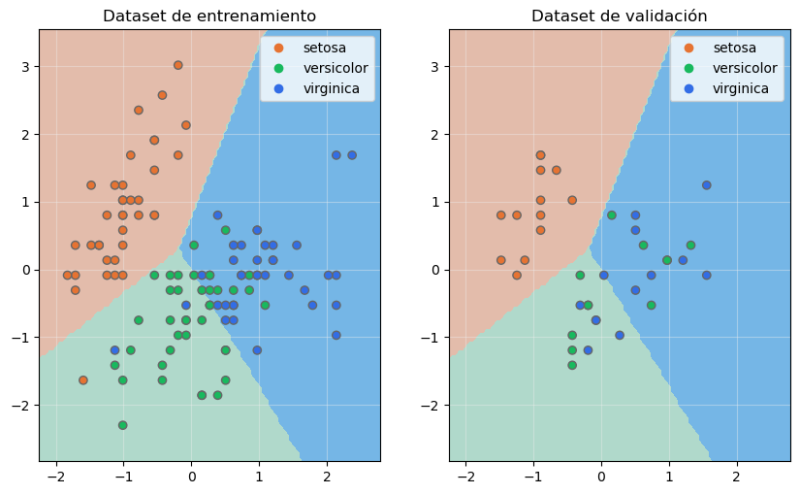
Vemos que las flores de la especie setosa del dataset de validación han sido correctamente clasificadas, aunque esta métrica ha sido mucho peor si nos fijamos en las flores de las otras dos especies.