Repitamos este análisis aplicándolo a datos no linealmente separables, como los resultantes de filtrar el dataset Iris para quedarnos solo con las especies versicolor y virginica:
iris = sns.load_dataset("iris")
iris = iris[iris.species.isin(["versicolor", "virginica"])]
iris["label"] = iris.species.astype("category").cat.codes
iris = iris[iris.species.isin(["versicolor", "virginica"])]
iris["label"] = iris.species.astype("category").cat.codes
y = iris.label.values
X = iris[["sepal_length", "sepal_width"]].values
X = iris[["sepal_length", "sepal_width"]].values
Visualicemos los datos antes de entrenar los modelos:
show_boundaries(None, X, None, y, None, iris.species.unique())
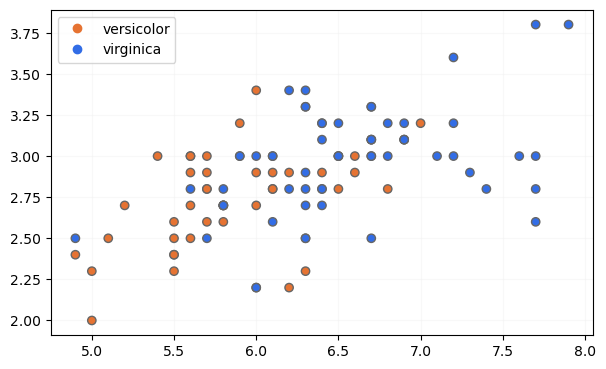
Claramente, se trata de datos no linealmente separables y con un porcentaje de solape bastante elevado.